DNA甲基化是表观遗传学(Epigenetics)的重要组成部分,在维持正常细胞功能、遗传印记、胚胎发育以及人类肿瘤发生中起着重要的作用。Bisulfite处理能够将基因组中未甲基化的胞嘧啶与甲基化的胞嘧啶区分开来,因此成为表观遗传学研究的经典实验方法。Bisulfite处理与高通量测序技术相结合的测序方法—Bisulfite Sequencing能够绘制单碱基分辨率的DNA甲基化图谱。特定物种的高精确度甲基化修饰模式的分析,为广泛应用于细胞分化、组织发育等基础机制研究,以及动植物育种、人类健康与疾病研究奠定基础。将样本DNA用亚硫酸氢钠处理,未甲基化的胞嘧啶被转变为尿嘧啶,而甲基化的胞嘧啶(5mC)则不受影响;而在PCR扩增反应中,尿嘧啶会被DNA聚合酶识别为胸腺嘧啶;最后采用特定的算法将测序所得reads与基因组进行比对就可获知样本DNA的甲基化情况。
信息分析
1、Bisulfite-seq数据产出统计: 数据产出总量;C的测序深度统计;基因组覆盖度统计,全基因组甲基化率统计;
2、计算C碱基的甲基化水平
3、全基因组甲基化水平数据分布趋势
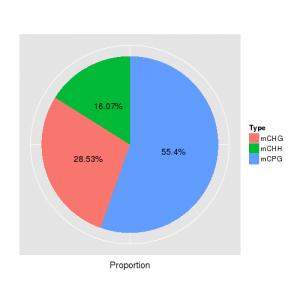
a)甲基化C碱基中CG, CHG 与CHH的分布比例
不同分布类型的甲基化C位点在不同物种基因组中出现比例不同,因此,各类型mC ( mCG、mCHG和mCHH ) 的位点数目,及其在全部mC的位点中所占的比例 ( 例:mCHG所占比例= mCHG数目/mC的总数 ),在一定程度上反映了特定物种的全基因组甲基化图谱的特征。
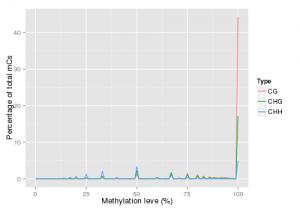
b)CG、CHG和CHH中的所有C的甲基化水平
不同分布类型的C 碱基( CG、CHG和CHH ),其甲基化水平在不同物种间,甚至同一物种不同细胞类型间都存在差异。此图统计每种类型 ( CG、CHG和CHH ) 甲基化的C甲基化水平的分布。
c)统计不同基因区域内CG、CHG和CHH中C的甲基化水平
对于在不同基因区域CG、CHG和CHH中的C,根据如下的公式统计其平均甲基化水平:平均甲基化水平=100*支持甲基化的reads的数目/总的reads的数目。(如:CG平均甲基化水平=100* 支持CG甲基化的reads/(支持CG甲基化的reads+支持CG非甲基化的reads)
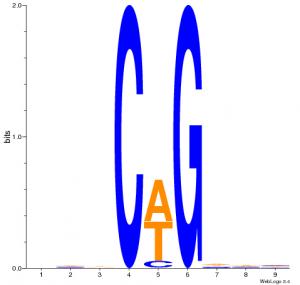
d)CHG,CHH中甲基化C附近的9bp序列的序列特征分析
分析非CG位点的甲基化的C附近碱基的分布情况,统计不同甲基化模式(CHG和CHH)出现的概率。图形横轴(x 轴)表示碱基位置,其中第四位上为用于分析C 碱基。纵轴(y 轴)为熵值(0 为最小值,表示四种碱基比例均匀,都为25%,2 为最大值,表示四种碱基分布最不均匀,即只有一种特定碱基出现,如第四位的C 与第六位的G)。
4、全基因组甲基化图谱
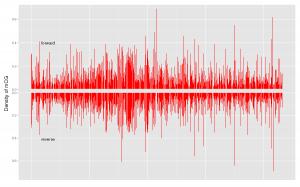
4.1染色体的甲基化分布
选取一条染色体,从染色体水平来描述甲基化C碱基的的分布情况。通过划窗口的方法来计算每个窗口的甲基化水平,下图窗口大小为10k。
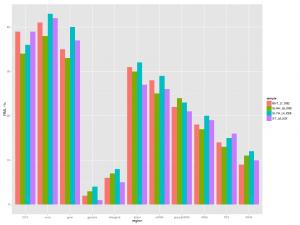
4.2不同基因组区域的甲基化分布特征
不同的基因组区域具有各自不同的生物学功能,下图中以柱形图的形式表示甲基化分布情况,有助于进一步了解这些区域的甲基化图谱特征。
5、差异性甲基化区域(DMRs)分析
比较两组样品间甲基化水平的差异对人类疾病研究、生长发育研究以及生物学的基础研究都具有重要的意义。这两组样品可以是不同类型的个体,可以是同一个体的不同组织,也可以是同一组织在不同时间点的取样。
5.1基于methyKit的差异甲基化(DMRs)分析
5.2基于BSmooth的差异甲基化(DMRs)分析
5.3DMRs相关基因的GO聚类分析
Gene Ontology(简称GO)是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词(controlled vocabulary)来全面描述生物体中基因和基因产物的属性。GO总共有三个ontology(本体),分别描述基因的分子功能(MF: molecular function)、所处的细胞位置(CC: cellular component)、参与的生物过程(BP: biological process)。

1. Heyn H, Vidal E, et al., Whole-genome bisulfite DNA sequencing of a DNMT3B mutant patient. Epigenetics. 2012;7(6):542-50.
2. Ehsan Habibi, at el. Whole-Genome Bisulfite Sequencing of Two Distinct Interconvertible DNA Methylomes of Mouse Embryonic Stem Cells. Cell Stem Cell 3(13), 360-369(2013).
3. Zhong S, Fei Z, Chen Y R, et al. Single-base resolution methylomes of tomato fruit development reveal epigenome modifications associated with ripening[J]. Nature biotechnology, 2013, 31(2): 154-159.
4. Kobayashi H, Sakurai T, Miura F, et al. High-resolution DNA methylome analysis of primordial germ cells identifies gender-specific reprogramming in mice[J]. Genome research, 2013, 23(4): 616-627.
1. Heyn H, Vidal E, et al., Whole-genome bisulfite DNA sequencing of a DNMT3B mutant patient. Epigenetics. 2012;7(6):542-50.
2. Ehsan Habibi, at el. Whole-Genome Bisulfite Sequencing of Two Distinct Interconvertible DNA Methylomes of Mouse Embryonic Stem Cells. Cell Stem Cell 3(13), 360-369(2013).
3. Zhong S, Fei Z, Chen Y R, et al. Single-base resolution methylomes of tomato fruit development reveal epigenome modifications associated with ripening[J]. Nature biotechnology, 2013, 31(2): 154-159.
4. Kobayashi H, Sakurai T, Miura F, et al. High-resolution DNA methylome analysis of primordial germ cells identifies gender-specific reprogramming in mice[J]. Genome research, 2013, 23(4): 616-627.