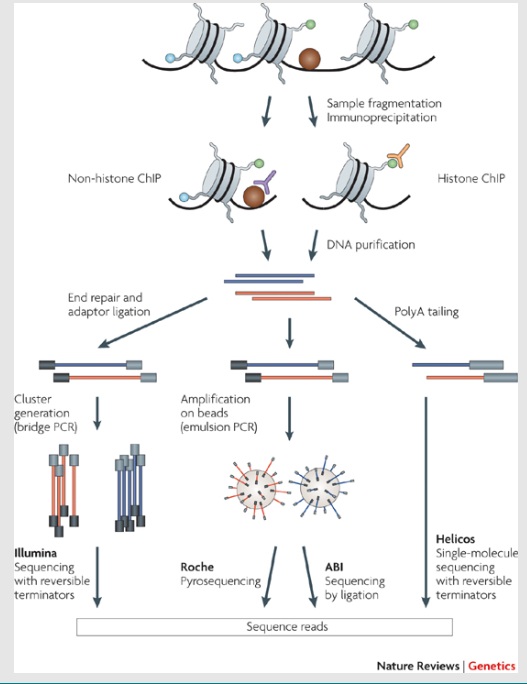
转录因子以及其他染色质相关蛋白对表观遗传有重要的影响。染色质免疫沉淀测序 (ChIP-sequencing,简称为ChIP-seq)是一种用于分析蛋白质与DNA交互作用的研究方法。该技术将染色质免疫沉淀 (Chromatin Immunoprecipitation, ChIP)与大规模并行DNA测序结合起来以鉴定DNA与相关蛋白结合的部位,可用于精确定位任意感兴趣的蛋白在全基因组上的结合位点,如广泛的应用于组 蛋白特异性修饰位点、转录因子结合位点等的研究。ChIP-Seq采用特异抗体对目的蛋白进行免疫沉淀,分离与目的蛋白结合的基因组DNA片段,对其进行纯化和文库构建,再通过高通量测序的方法,在全基 因组范围内寻找目的蛋白的DNA结合位点,从而获得全基因组范围内与组蛋白、转录因子等互作的DNA片段信息。
1、原始数据产出统计;
2、与基因组比对(需甲方提供参考基因组信息,如hg19);
3、比上的Reads在基因组各区域的分布;
4、Reads相对TSS(transcript start site)、TTS(transcript terminal site)的depth分布;
5、全基因组Peak Calling;
6、基因组上检测到的Peak统计分布(峰数目,峰间距,峰宽度等);
7、每个样本的检测到的Peak在基因组各区域的分布情况;
8、提供与样本Peaks相关的基因列表;
9、Peaks相关基因Gene Ontology功能分类;
10、Case 相对control的差异peaks及其相关基因差异统计;
7、每个样本的检测到的Peak在基因组各区域的分布情况;
8、提供与样本Peaks相关的基因列表;
9、Peaks相关基因Gene Ontology功能分类;
10、Case 相对control的差异 peaks及其相关基因差异统计;
1、需提供至少2ng 样品起始量的ChIP-seq建库产品,要求ChIP片段长度200-500bp,为保证实验质量及延续性,请一次提供至少2次样本制备的量;
2、样品浓度和纯度:基因组DNA样品浓度>0.5ng/μl;OD260/280介于1.8-2.0,无肉眼可见污染;
3、样品保存:请选择乙醇、纯水进行保存,并在样品信息单中注明;
4、样品运输:请将样品置于1.5ml管中,并用封口膜封好。
研究背景
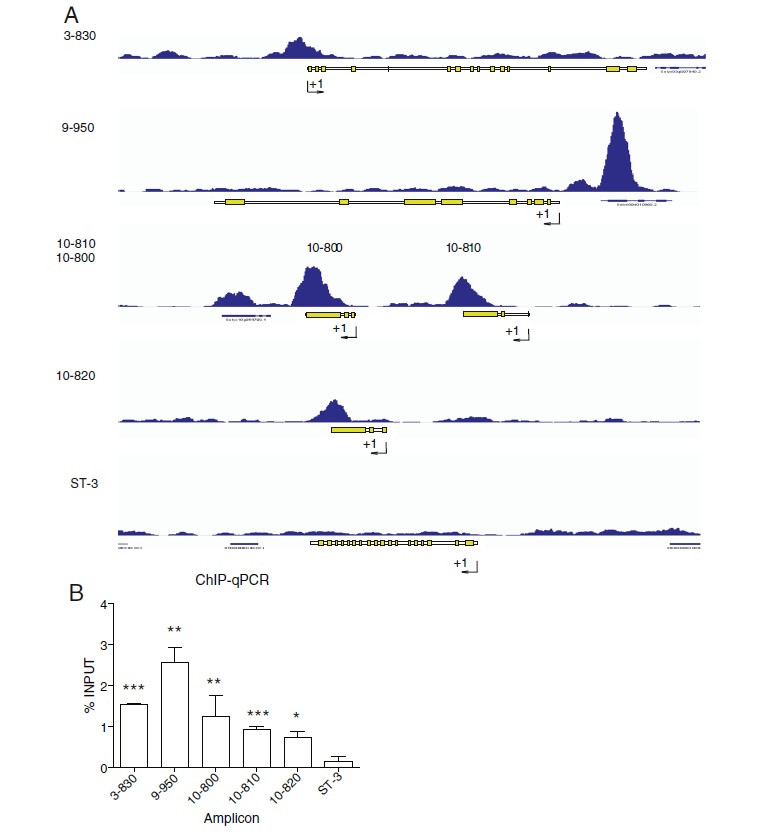
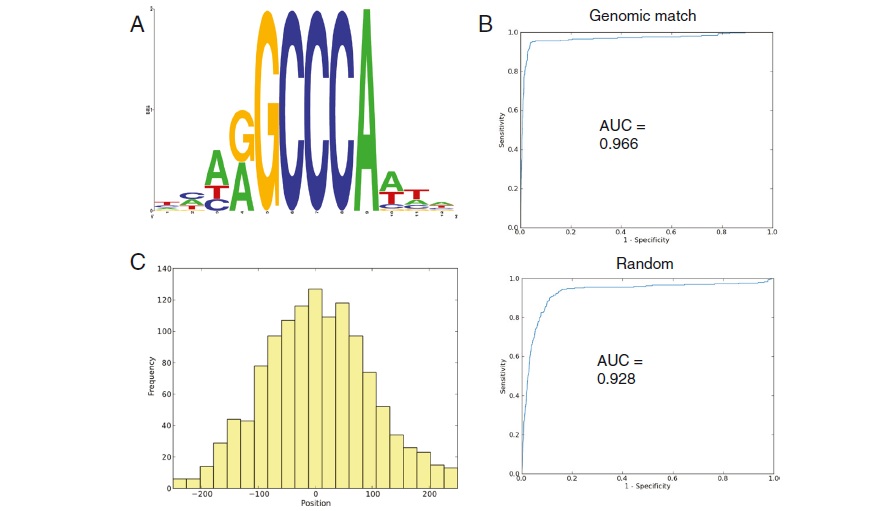
鉴定转录因子的靶基因,对于揭示生物体中的调控网络非常重要。干旱是植物常见逆境胁迫的一种,本研究目的是精确解析植物干旱耐受相关转录因子ASR1调控的基因座位点。ASR1是植物界(拟南芥除外)独有的调节因子,可缓解缺水引起的胁迫。但是ASR1的靶基因仍然不清楚,本文检测西红柿中与ASR1蛋白虎作的特殊基因座位点。
研究方法
采用高质量的anti-ASR1抗体,通过CHIP沉淀西红柿叶片中的DNA、序列,然后进行高通量的DNA测序分析。
研究结果
1.鉴定到新的靶基因库,其中一些基因明确参与干旱胁迫响应;
2.ASR1富集到的一些基因,编码细胞壁合成相关蛋白和水通道蛋白;
3.鉴定到超强一致性的ASR1互作DNA元件。
参考文献:
Ricardi M M, González R M, Zhong S, et al. Genome-wide data (ChIP-seq) enabled identification of cell wall-related and aquaporin genes as targets of tomato ASR1, a drought stress-responsive transcription factor[J]. BMC plant biology, 2014, 14(1): 29.